|
|
2月5日凌晨更新了SEIRS模型。
<hr/>今天是2020年1月27日。根据国家卫生健康委的数据,截至2020年1月26日24时,中国30个省(区、市)累积报告确诊病例2744例,重症病例461例,累计死亡病例80例,累计治愈出院51例,疑似病例5794例,累计追踪密切接触者32799人,当日解除医学观察583人,现有30453人正在接受医学观察。
随着1月23日武汉市宣布全面封城后,各地确诊病例不断出现,2020年的新年因为新型冠状病毒(2019-nCoV)的传播而黯然失色。
本文尝试使用python对经典传染病模型进行实现,因传染病模型研究属于传染病动力学研究方向,不是本人的工作范围,因此,本人只是将模型中的微分方程,用Python进行实现,想起到抛砖引玉的效果。
(具体各个模型的理论细节,请移步其他文章)
模型一:SI-Model
import scipy.integrate as spi
import numpy as np
import matplotlib.pyplot as plt
# N为人群总数
N = 10000
# β为传染率系数
beta = 0.25
# gamma为恢复率系数
gamma = 0
# I_0为感染者的初始人数
I_0 = 1
# S_0为易感者的初始人数
S_0 = N - I_0
# T为传播时间
T = 150
# INI为初始状态下的数组
INI = (S_0,I_0)
def funcSI(inivalue,_):
Y = np.zeros(2)
X = inivalue
# 易感个体变化
Y[0] = - (beta * X[0] * X[1]) / N + gamma * X[1]
# 感染个体变化
Y[1] = (beta * X[0] * X[1]) / N - gamma * X[1]
return Y
T_range = np.arange(0,T + 1)
RES = spi.odeint(funcSI,INI,T_range)
plt.plot(RES[:,0],color = &#39;darkblue&#39;,label = &#39;Susceptible&#39;,marker = &#39;.&#39;)
plt.plot(RES[:,1],color = &#39;red&#39;,label = &#39;Infection&#39;,marker = &#39;.&#39;)
plt.title(&#39;SI Model&#39;)
plt.legend()
plt.xlabel(&#39;Day&#39;)
plt.ylabel(&#39;Number&#39;)
plt.show()

模型二:SIS-Model
import scipy.integrate as spi
import numpy as np
import matplotlib.pyplot as plt
# N为人群总数
N = 10000
# β为传染率系数
beta = 0.25
# gamma为恢复率系数
gamma = 0.05
# I_0为感染者的初始人数
I_0 = 1
# S_0为易感者的初始人数
S_0 = N - I_0
# T为传播时间
T = 150
# INI为初始状态下的数组
INI = (S_0,I_0)
def funcSIS(inivalue,_):
Y = np.zeros(2)
X = inivalue
# 易感个体变化
Y[0] = - (beta * X[0]) / N * X[1] + gamma * X[1]
# 感染个体变化
Y[1] = (beta * X[0] * X[1]) / N - gamma * X[1]
return Y
T_range = np.arange(0,T + 1)
RES = spi.odeint(funcSIS,INI,T_range)
plt.plot(RES[:,0],color = &#39;darkblue&#39;,label = &#39;Susceptible&#39;,marker = &#39;.&#39;)
plt.plot(RES[:,1],color = &#39;red&#39;,label = &#39;Infection&#39;,marker = &#39;.&#39;)
plt.title(&#39;SIS Model&#39;)
plt.legend()
plt.xlabel(&#39;Day&#39;)
plt.ylabel(&#39;Number&#39;)
plt.show()
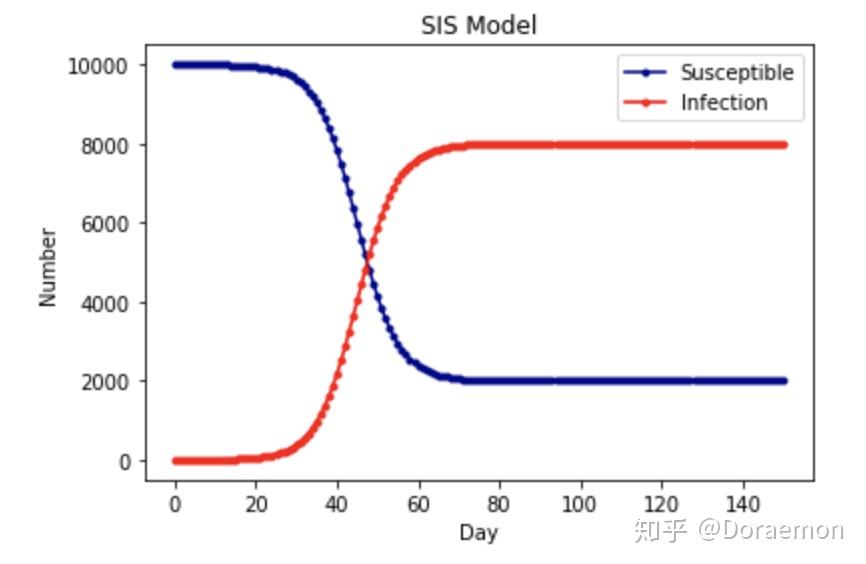
模型三:SIR-Model
import scipy.integrate as spi
import numpy as np
import matplotlib.pyplot as plt
# N为人群总数
N = 10000
# β为传染率系数
beta = 0.25
# gamma为恢复率系数
gamma = 0.05
# I_0为感染者的初始人数
I_0 = 1
# R_0为治愈者的初始人数
R_0 = 0
# S_0为易感者的初始人数
S_0 = N - I_0 - R_0
# T为传播时间
T = 150
# INI为初始状态下的数组
INI = (S_0,I_0,R_0)
def funcSIR(inivalue,_):
Y = np.zeros(3)
X = inivalue
# 易感个体变化
Y[0] = - (beta * X[0] * X[1]) / N
# 感染个体变化
Y[1] = (beta * X[0] * X[1]) / N - gamma * X[1]
# 治愈个体变化
Y[2] = gamma * X[1]
return Y
T_range = np.arange(0,T + 1)
RES = spi.odeint(funcSIR,INI,T_range)
plt.plot(RES[:,0],color = &#39;darkblue&#39;,label = &#39;Susceptible&#39;,marker = &#39;.&#39;)
plt.plot(RES[:,1],color = &#39;red&#39;,label = &#39;Infection&#39;,marker = &#39;.&#39;)
plt.plot(RES[:,2],color = &#39;green&#39;,label = &#39;Recovery&#39;,marker = &#39;.&#39;)
plt.title(&#39;SIR Model&#39;)
plt.legend()
plt.xlabel(&#39;Day&#39;)
plt.ylabel(&#39;Number&#39;)
plt.show()
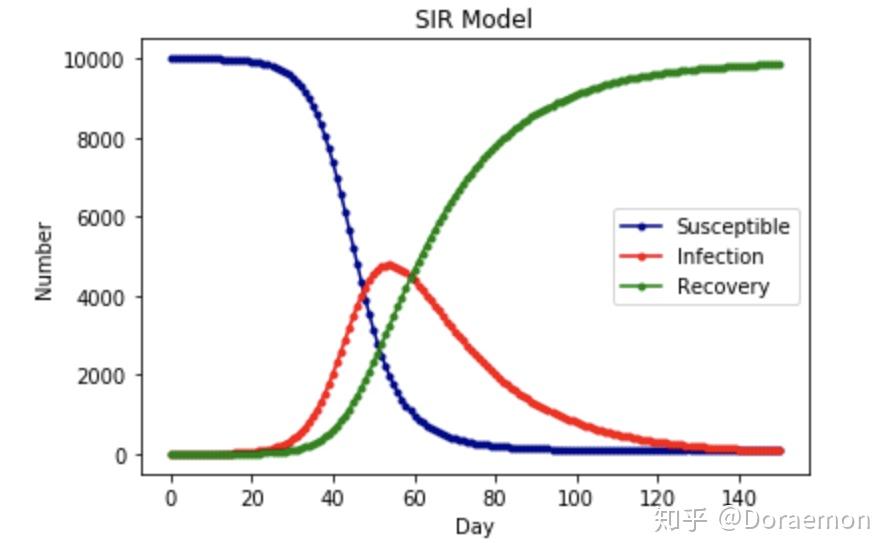
模型四:SIRS-Model
import scipy.integrate as spi
import numpy as np
import matplotlib.pyplot as plt
# N为人群总数
N = 10000
# β为传染率系数
beta = 0.25
# gamma为恢复率系数
gamma = 0.05
# Ts为抗体持续时间
Ts = 7
# I_0为感染者的初始人数
I_0 = 1
# R_0为治愈者的初始人数
R_0 = 0
# S_0为易感者的初始人数
S_0 = N - I_0 - R_0
# T为传播时间
T = 150
# INI为初始状态下的数组
INI = (S_0,I_0,R_0)
def funcSIRS(inivalue,_):
Y = np.zeros(3)
X = inivalue
# 易感个体变化
Y[0] = - (beta * X[0] * X[1]) / N + X[2] / Ts
# 感染个体变化
Y[1] = (beta * X[0] * X[1]) / N - gamma * X[1]
# 治愈个体变化
Y[2] = gamma * X[1] - X[2] / Ts
return Y
T_range = np.arange(0,T + 1)
RES = spi.odeint(funcSIRS,INI,T_range)
plt.plot(RES[:,0],color = &#39;darkblue&#39;,label = &#39;Susceptible&#39;,marker = &#39;.&#39;)
plt.plot(RES[:,1],color = &#39;red&#39;,label = &#39;Infection&#39;,marker = &#39;.&#39;)
plt.plot(RES[:,2],color = &#39;green&#39;,label = &#39;Recovery&#39;,marker = &#39;.&#39;)
plt.title(&#39;SIRS Model&#39;)
plt.legend()
plt.xlabel(&#39;Day&#39;)
plt.ylabel(&#39;Number&#39;)
plt.show()
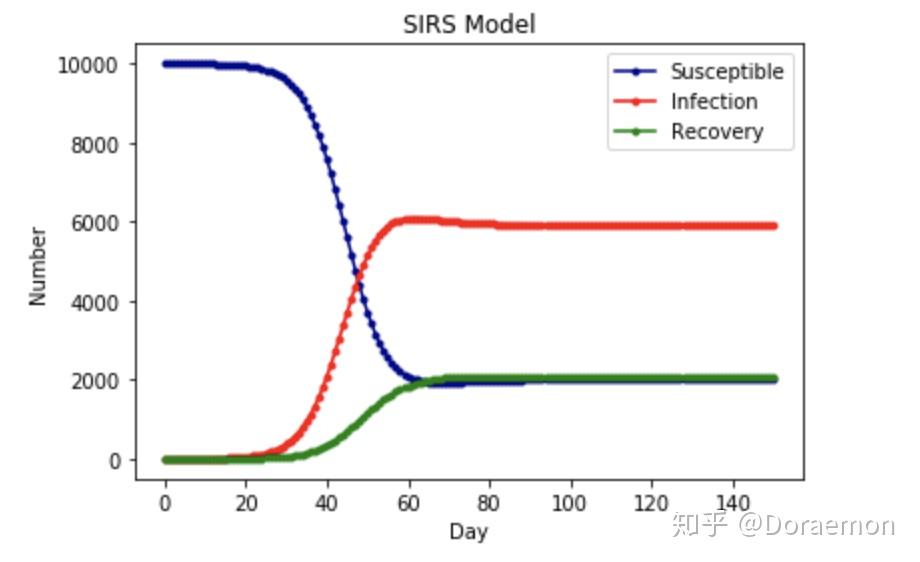
模型五:SEIR-Model
import scipy.integrate as spi
import numpy as np
import matplotlib.pyplot as plt
# N为人群总数
N = 10000
# β为传染率系数
beta = 0.6
# gamma为恢复率系数
gamma = 0.1
# Te为疾病潜伏期
Te = 14
# I_0为感染者的初始人数
I_0 = 1
# E_0为潜伏者的初始人数
E_0 = 0
# R_0为治愈者的初始人数
R_0 = 0
# S_0为易感者的初始人数
S_0 = N - I_0 - E_0 - R_0
# T为传播时间
T = 150
# INI为初始状态下的数组
INI = (S_0,E_0,I_0,R_0)
def funcSEIR(inivalue,_):
Y = np.zeros(4)
X = inivalue
# 易感个体变化
Y[0] = - (beta * X[0] * X[2]) / N
# 潜伏个体变化
Y[1] = (beta * X[0] * X[2]) / N - X[1] / Te
# 感染个体变化
Y[2] = X[1] / Te - gamma * X[2]
# 治愈个体变化
Y[3] = gamma * X[2]
return Y
T_range = np.arange(0,T + 1)
RES = spi.odeint(funcSEIR,INI,T_range)
plt.plot(RES[:,0],color = &#39;darkblue&#39;,label = &#39;Susceptible&#39;,marker = &#39;.&#39;)
plt.plot(RES[:,1],color = &#39;orange&#39;,label = &#39;Exposed&#39;,marker = &#39;.&#39;)
plt.plot(RES[:,2],color = &#39;red&#39;,label = &#39;Infection&#39;,marker = &#39;.&#39;)
plt.plot(RES[:,3],color = &#39;green&#39;,label = &#39;Recovery&#39;,marker = &#39;.&#39;)
plt.title(&#39;SEIR Model&#39;)
plt.legend()
plt.xlabel(&#39;Day&#39;)
plt.ylabel(&#39;Number&#39;)
plt.show()
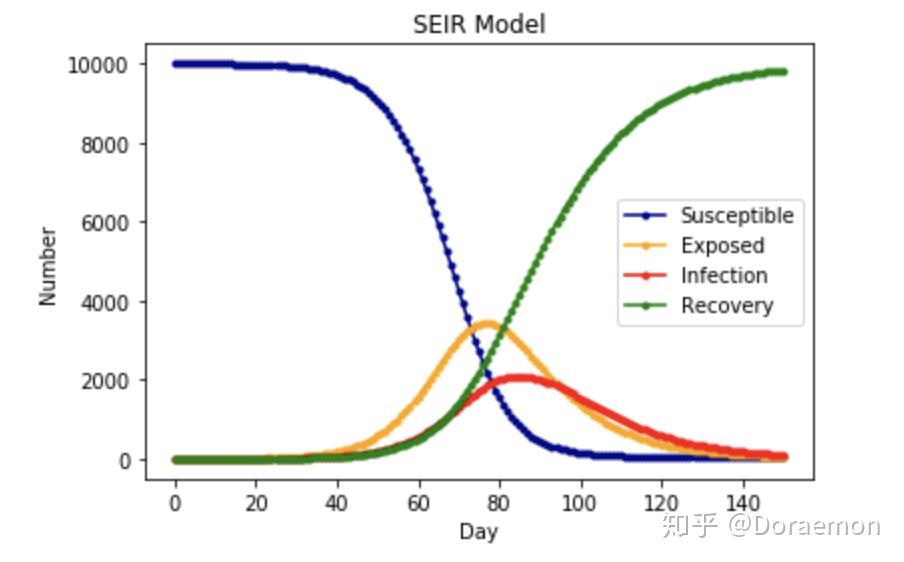
模型六:SEIRS-Model
import scipy.integrate as spi
import numpy as np
import matplotlib.pyplot as plt
# N为人群总数
N = 10000
# β为传染率系数
beta = 0.6
# gamma为恢复率系数
gamma = 0.1
# Ts为抗体持续时间
Ts = 7
# Te为疾病潜伏期
Te = 14
# I_0为感染者的初始人数
I_0 = 1
# E_0为潜伏者的初始人数
E_0 = 0
# R_0为治愈者的初始人数
R_0 = 0
# S_0为易感者的初始人数
S_0 = N - I_0 - E_0 - R_0
# T为传播时间
T = 150
# INI为初始状态下的数组
INI = (S_0,E_0,I_0,R_0)
def funcSEIRS(inivalue,_):
Y = np.zeros(4)
X = inivalue
# 易感个体变化
Y[0] = - (beta * X[0] * X[2]) / N + X[3] / Ts
# 潜伏个体变化
Y[1] = (beta * X[0] * X[2]) / N - X[1] / Te
# 感染个体变化
Y[2] = X[1] / Te - gamma * X[2]
# 治愈个体变化
Y[3] = gamma * X[2] - X[3] / Ts
return Y
T_range = np.arange(0,T + 1)
RES = spi.odeint(funcSEIRS,INI,T_range)
plt.plot(RES[:,0],color = &#39;darkblue&#39;,label = &#39;Susceptible&#39;,marker = &#39;.&#39;)
plt.plot(RES[:,1],color = &#39;orange&#39;,label = &#39;Exposed&#39;,marker = &#39;.&#39;)
plt.plot(RES[:,2],color = &#39;red&#39;,label = &#39;Infection&#39;,marker = &#39;.&#39;)
plt.plot(RES[:,3],color = &#39;green&#39;,label = &#39;Recovery&#39;,marker = &#39;.&#39;)
plt.title(&#39;SEIRS Model&#39;)
plt.legend()
plt.xlabel(&#39;Day&#39;)
plt.ylabel(&#39;Number&#39;)
plt.show()
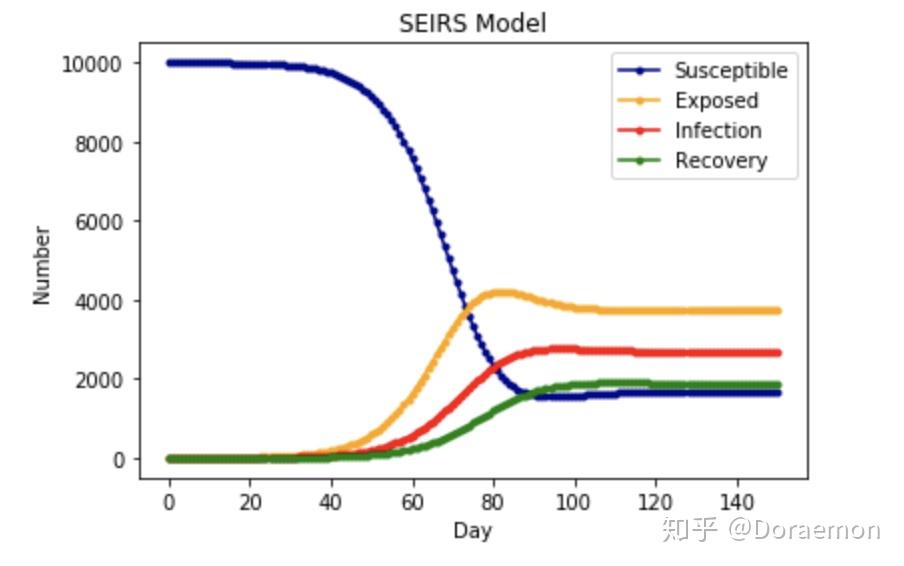
关于2019-nCoV的SEIR模型参数设置不具备参考性,所以做了删除处理。后续时间可能会更改重新放在这里,也可能更新其他模型(如考虑常量输出输入的SEIR模型、SQIR模型等。。。)
因本人以前从未研究过传染病动力学,只是一时感兴趣,所以不免存在问题,欢迎大家指正~
本文参考的博客链接:
欢迎关注~一个医学生(预防医学)的数据成长之路。。。 |
|